Différence entre le séquençage du génome entier et les puces à ADN
- 3209
- 50
- Mathilde Roux
Le séquençage de l'ADN est la détermination de la disposition des blocs de construction chimique appelés «nucléobases» dans toute la chaîne de molécule d'ADN. Ce décodage des séquences génétiques montre aux scientifiques les informations génétiques codées dans un segment d'ADN donné. Dans la double hélice de l'ADN, chacune des bases chimiques se lie toujours de la même manière, pour former des paires de bases.«L'adénine (a) se lie toujours à la thymine (t), et la cytosine (c) se lie toujours à la guanine (g). Cet appariement sous-tend le mécanisme par lequel les molécules d'ADN sont copiées pendant la division cellulaire. L'association est la base des méthodes utilisées pour le séquençage d'ADN. Il existe différentes techniques de séquençage, utilisées à différentes fins.
La capacité de l'ADN à coder les informations génétiques est énorme - le nombre possible de configurations d'un brin d'ADN composé de n nucléotides est 4n. Selon les objectifs de l'analyse, il peut être nécessaire de révéler l'ensemble du génome d'un individu ou d'identifier uniquement si certains gènes sont présents. Le processus par lequel la séquence d'ADN complète d'un génome est déterminée est appelée séquençage du génome entier. Le processus de détection de l'expression des gènes de certains gènes prédéfinis à l'aide de lames de microscope avec certaines séquences d'ADN ou gènes situées à des positions définies est appelée microréseau.

Qu'est-ce que le séquençage du génome entier?
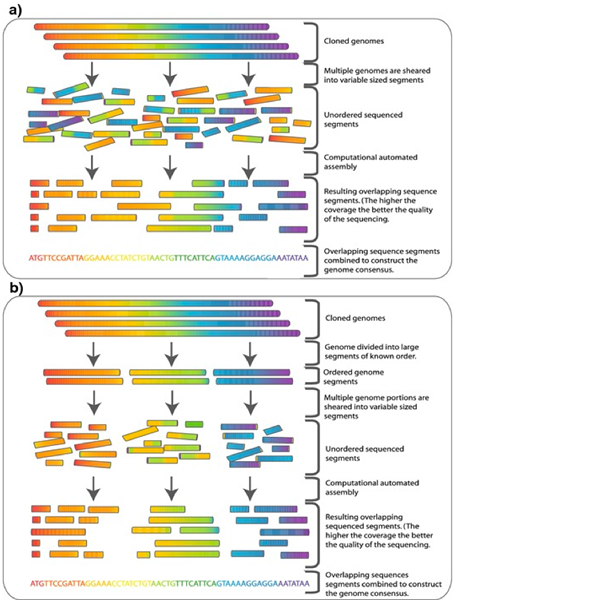
Le séquençage du génome entier est le processus par lequel la séquence d'ADN complète du génome humain, animal, végétal, bactérien ou viral est déterminée simultanément. Grâce au séquençage du génome entier, toutes les informations héréditaires d'un organisme vivant sont lues. Le résultat est une liste avec la séquence nucléotidique brute de l'ensemble haploïde de chromosomes. Le séquençage du génome entier est un outil puissant pour diagnostiquer les maladies génétiques rares. Cependant, pour comprendre la signification médicale ou biologique des informations obtenues, une analyse plus approfondie est nécessaire.
En 2003, le génome humain a été complètement déchiffré pour la première fois dans un programme de recherche international lancé en 1990 appelé Human Genome Project (HGP). Le génome humain contient environ 3 milliards de paires de bases et est l'ensemble complet des «instructions» de codage pour la création et la reproduction de chaque organisme. Le séquençage du génome entier génère une énorme quantité de données et a besoin de capacité de stockage et de puissance de calcul pertinente.
Qu'est-ce que les microréseaux?
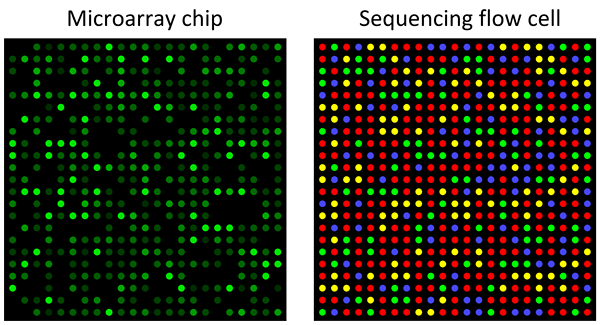
Les microréseaux sont la technologie de laboratoire pour la détection de l'expression des gènes, une bibliothèque particulière d'échantillons d'acide nucléique synthétiques immobilisés, disposés spatialement sur une base solide. Il utilise une diapositive de microscope avec certaines séquences d'ADN ou des gènes situés à des positions définies. L'utilisation de l'hybridation dans des séquences complémentaires, cette technologie aide à profiter et à étudier simultanément le transcriptome (i.e. L'ensemble de toutes les molécules d'ARN exprimées dans une cellule).
La technologie ADN ADN a été développée au milieu des années 1990. La base de cette technologie est l'hybridation entre l'ADNc inversé transcrit d'un échantillon de cellule à une diapositive avec une sonde d'ADN complémentaire attachée.
Initialement, les microréseaux étaient composés de papier filtre avec des ADNc immobilisés. Depuis 1995, ils sont constitués de sondes synthétiques courtes attachées à des endroits spécifiquement désignés sur la base solide (silicium ou verre).
Les microréseaux d'ADN sont utilisés dans le diagnostic de la maladie pour la détection de certains gènes pertinents par la maladie ou de leurs biomarqueurs. Ils sont également utilisés pour l'analyse des mutations, la détection des anomalies chromosomiques, le dépistage des polymorphismes un seul nucléotide et la détermination de la modification post-traductionnelle.
Le principe principal derrière cette méthode est la capacité des séquences d'ADN à s'associer à des séquences complémentaires via des liaisons hydrogène. Le nombre élevé de paires de bases complémentaires assure un lien fort. Les séquences non liées non spécifiquement sont lavées tandis que les fortement liés restent attachés au microréseau.
Différence entre le séquençage du génome entier et les puces à ADN
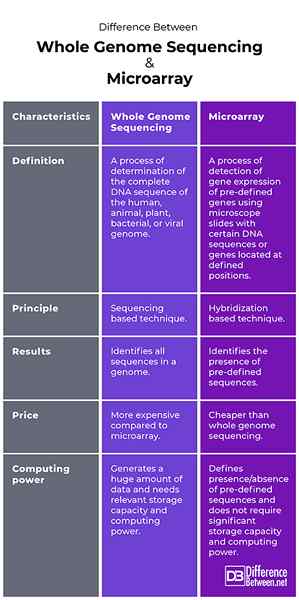
Définition
Séquençage du génome entier: Le séquençage du génome entier est un processus de détermination de la séquence d'ADN complète du génome humain, animal, végétal, bactérien ou viral.
ADN: Le processus de détection de l'expression des gènes des gènes prédéfinis à l'aide de lames de microscope avec certaines séquences d'ADN ou gènes situées à des positions définies est appelée microréseau.
Principe
Séquençage du génome entier: Le séquençage du génome entier est une technique basée sur le séquençage.
ADN: ADN est une technique basée sur l'hybridation.
Résultats
Séquençage du génome entier: Le séquençage entièrementgeme identifie toutes les séquences dans un génome.
ADN: ADN identifie la présence de séquences prédéfinies.
Prix
Séquençage du génome entier: Le séquençage du génome entier est plus cher par rapport aux puces à ADN.
ADN: ADN est moins cher que le séquençage du génome entier.
Puissance de calcul
Séquençage du génome entier: Le séquençage du génome entier génère une énorme quantité de données et a besoin de capacité de stockage et de puissance de calcul pertinente.
ADN: Un microréseau définit la présence / l'absence de séquences prédéfinies et ne nécessite pas de capacité de stockage et de puissance de calcul significative.
Différence entre le séquençage du génome entier et les puces à ADN
Résumé:
- Chaque molécule d'ADN est un chromosome séparé emballé, et toutes les informations génétiques contenues dans l'ensemble haploïde de chromosomes de chaque organisme sont appelés génomes.
- Selon les objectifs de l'analyse, il peut être nécessaire de révéler l'ensemble du génome d'un individu ou d'identifier uniquement si certains gènes sont présents.
- Le processus par lequel la séquence d'ADN complète d'un génome est déterminée est appelée séquençage du génome entier.
- Le processus de détection de l'expression des gènes des gènes prédéfinis à l'aide de lames de microscope avec certaines séquences d'ADN ou gènes situées à des positions définies est appelée microréseau.
- Le séquençage du génome entier est une technique basée sur le séquençage, tandis que les microréseaux sont une technique basée sur l'hybridation.
- Le séquençage du génome entier identifie toutes les séquences dans un génome, tandis que le microréseau identifie la présence d'un ensemble prédéfini de séquences.
- Le séquençage du génome entier est plus cher que les puces à ADN.
- Le séquençage du génome entier génère une énorme quantité de données et a besoin de capacité de stockage et de puissance de calcul pertinente. Un microréseau définit la présence / l'absence de séquences prédéfinies et ne nécessite pas de capacité de stockage et de puissance de calcul significative.

